基于PCA与LDA的数据降维实践
描述
数据降维(Dimension Reduction)是降低数据冗余、消除噪音数据的干扰、提取有效特征、提升模型的效率和准确性的有效途径, PCA(主成分分析)和LDA(线性判别分析)是机器学习和数据分析中两种常用的经典降维算法。
本任务通过两个降维案例熟悉PCA和LDA降维的原理、区别及调用方法。
源码下载
环境
-
操作系统:Windows 10、Ubuntu18.04
-
工具软件:Anaconda3 2019、Python3.7
-
硬件环境:无特殊要求
-
依赖库列表
matplotlib 3.3.4 scikit-learn 0.24.2
分析
任务1、基于PCA算法实现鸢尾花数据集降维,涉及下列三个环节:
A)加载鸢尾花(Iris)数据并进行降维
B)降维后的数据可视化
C)使用K-NN算法进行分类,对比降维前后的分类准确性
任务2、基于LDA算法实现红酒数据集降维,涉及以下四个环节:
A)加载红酒数据集
B)使用PCA和LDA两种算法对数据进行降维
C)降维结果可视化
D)降维前后的分类准确性对比
实施
1、基于PCA算法实现鸢尾花数据集降维
鸢尾花数据原有四个特征维度,运用PCA算法将特征维度降为两个,之后进行可视化并运用K-NN算法进行分类,对比降维前后的分类准确性(数据降维的目的之一是提升模型的准确性)。
1.1 加载鸢尾花特征数据,并使用PCA算法降维
import matplotlib.pyplot as plt
from sklearn.decomposition import PCA
from sklearn.datasets import load_iris
from sklearn.neighbors import KNeighborsClassifier# 加载鸢尾花数据集
iris= load_iris()
data = iris.data # 特征数据
target = iris.target # 标签数据
print(data.shape) # 查看数据维度(150, 4)# PCA降维
pca = PCA(n_components = 2).fit(data) # 利用PCA算法降成2维
new_data = pca.transform(data)
print(new_data.shape) # 查看数据维度(150,2)
结果如下:
(150, 4)
(150, 2)
可以看到,鸢尾花数据由四维(四个特征)降为两维度。
1.2 数据可视化,并使用K-NN算法对比降维前后的分类准确性
# 降维后的数据集可视化
plt.title('Iris dimensions reduction: 4 to 2')
plt.scatter(new_data[:, 0], new_data[:, 1], c=target)
plt.show()# 使用KNN算法对比降维前后分类的准确性
model = KNeighborsClassifier(3)
score = model.fit(data, target).score(data, target)
print('4-dims:', score)
score = model.fit(new_data, target).score(new_data, target)
print('2-dims:', score)
输出结果:
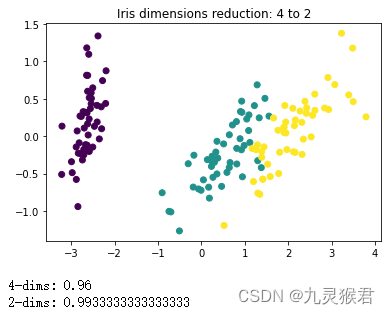
结果分析:
数据从4维降到2维后,可以很方便地进行可视化。从散点图中直观地看,降维后的数据较好地保留了原数据的分布信息。另外可以看到,降维后的KNN分类模型准确性有所提升,这也是数据降维的目的之一。
2、基于LDA算法实现红酒数据集降维
红酒数据集(Wine)有13个特征(即13个维度),我们分别使用PCA和LDA算法对数据集进行降维(降成2维),之后使用逻辑回归(LogisticRegression)分别在LDA算法降维前后的数据集上建立分类模型,对比同一种模型在数据集降维前后的准确性,直观感受数据降维对模型准确性的影响。
2.1 加载红酒数据集
import matplotlib.pyplot as plt
from sklearn.decomposition import PCA
from sklearn.discriminant_analysis import LinearDiscriminantAnalysis
from sklearn.datasets import load_wine# 加载红酒数据集
wine= load_wine()
data = wine.data
target = wine.target
print(data.shape) # 查看数据维度
2.2 分别使用LDA和PCA算法进行降维
# PCA降维(无类别)
pca = PCA(n_components = 2).fit(data) # 利用PCA算法降成2维
data_pca = pca.transform(data) # 降维转换
print('PCA:', data_pca.shape) # 查看数据维度# LDA降维(有类别,考虑样本标签)
lda = LinearDiscriminantAnalysis(n_components=2).fit(data, target)
data_lda = lda.transform(data)
print('LDA:', data_lda.shape)
结果如下:
(178, 13)
PCA: (178, 2)
LDA: (178, 2)
可以看到,两种算法都将红酒数据集由13维降成2维。
2.3 降维结果可视化
数据降到2维后,可以很方便地用散点图进行可视化,下面分别将两种算法降维后的红酒数据集进行可视化,对比其分布情况。
# LDA算法更适合有标签数据的降维
# 下面将两种方法降维后的数据进行可视化
fig = plt.figure(figsize=(12, 4)) # 生成画板# PCA降维结果
ax1 = fig.add_subplot(1, 2, 1) # 添加子图1
ax1.set_title('PCA')
ax1.scatter(data_pca[:, 0], data_pca[:, 1], c=target)# LDA降维结果
ax2 = fig.add_subplot(1, 2, 2) # 添加子图2
ax2.set_title('LDA')
ax2.scatter(data_lda[:, 0], data_lda[:, 1], c=target)plt.show() # 显示图像
显示结果:

可以看到,LDA降维因为考虑到了样本的类别标签信息,降维后的数据分布能够较好地将类型分开。
2.4 LDA降维前后的分类准确性对比
使用逻辑回归算法,对LDA降维前后的数据集建立分类模型,对比其准确性。
from sklearn.model_selection import train_test_split
from sklearn.linear_model import LogisticRegression# 1、使用逻辑回归模型,在降维前的数据集上训练并评估
X_train, X_test, y_train, y_test = train_test_split(data, target, random_state=0)model = LogisticRegression().fit(X_train, y_train)
score = model.score(X_test, y_test) # 在测试集上评估分类准确性
print(score)# 2、在LDA降维后的数据集上训练并评估
X_train, X_test, y_train, y_test = train_test_split(data_lda, target, random_state=0)
model = LogisticRegression().fit(X_train, y_train)
score = model.score(X_test, y_test) # 在测试集上评估分类准确性
print(score)
结果如下:
0.9333333333333333
1.0
可以看到,使用LDA降维后的数据建模,分类准确性有所提升。
本文链接:https://my.lmcjl.com/post/5342.html

4 评论